Diferencia entre revisiones de «Cambios genéticos como fuente de variación.»
| Línea 46: | Línea 46: | ||
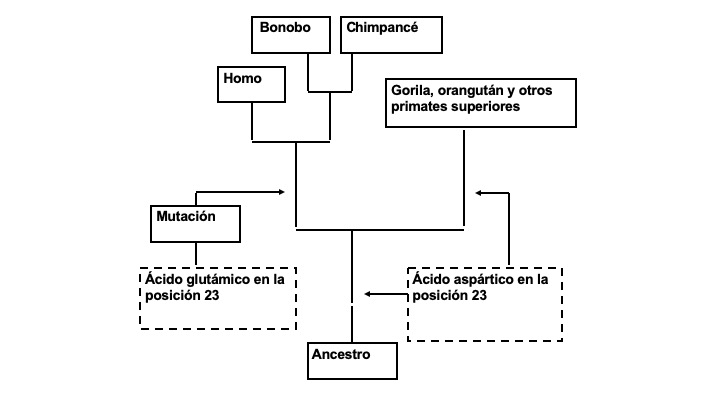
<center>Figura 3. Mutación que cambió el ácido aspártico por ácido glutámico en el ancestro de chimpancés y humanos.</center> | <center>Figura 3. Mutación que cambió el ácido aspártico por ácido glutámico en el ancestro de chimpancés y humanos.</center> | ||
| + | |||
| + | <center>[[Archivo:alfa y beta en animales.jpg]]</center> | ||
Revisión del 21:28 16 may 2019
Lectura: Cambios genéticos como fuente de variación
La gran diversidad biológica que se puede estudiar en tres niveles: genético, de especies y de ecosistemas, es el resultado del proceso evolutivo, cuyos cimientos son la variabilidad genética existente entre especies y entre individuos de una misma especie sujetos a la selección natural. Si estudiamos las causas de la diversidad, primero tenemos que darnos cuenta de las sutiles diferencias existentes entre individuos de la misma especie. Estas diferencias se deben principalmente a que los individuos de una población, frecuentemente presentan diferentes combinaciones de las versiones existentes de un mismo gen (alelos).
Las diferencias genéticas dentro de una misma especie pueden llevar a pequeños grupos de la población, ya sea en ambientes diferentes o incluso en un mismo ambiente a la acumulación de ciertas características (alelos) que los pueden hacer lo suficientemente diferentes como para aislarlos reproductivamente de la población original y formar una variedad o incluso una nueva especie. Desde luego que la fuente primaria de variación es la mutación, y está sujeta a la selección natural y a la deriva génica.
En esta lectura abordaremos algunos aspectos sobresalientes con relación al ADN y ARN desde la perspectiva de la diversidad genética, para lo cual tendrás que hacer un repaso sobre la estructura y función de los ácidos nucleicos que estudiaste en tu curso de Biología I, así como de la síntesis de proteínas y la expresión génica. La función del ADN y ARN se puede comparar a la capacidad de escribir, leer y seguir instrucciones a nivel molecular. Lo deseable es que en clase se haga un repaso de lo visto en Biología I para abordar el tema sin mayores dificultades y esto lo puedes hacer con la ayuda de tu profesor.
Los seres vivos utilizan un lenguaje especial que contiene la información necesaria para la síntesis de proteínas y el funcionamiento general de un individuo, que llamamos código genético y que funciona más o menos de la siguiente manera: La célula transcribe “escribe” su información genética en forma codificada en las moléculas de ARNm a partir de la información del ADN y las envía al citoplasma, en donde se encuentran los ribosomas, que de acuerdo a las instrucciones codificadas en el ARNm ensamblan los aminoácidos en una secuencia específica indicada por el mismo ARN. Este proceso es el equivalente a la lectura del mensaje para la síntesis de proteínas y que se traduce en una proteína. Esto último es la capacidad de seguir instrucciones. Una célula que no pueda realizar lo anterior está condenada a morir.
Como recordarás, el ADN de todos los seres vivos está constituido por los mismos monómeros.
Bases nitrogenadas: adenina, timina, guanina y citosina. Carbohidrato: desoxirribosa. Ácido fosfórico.
Las diferencias entre individuos y entre especies se deben al ordenamiento (secuencia) de las bases nitrogenadas en el ADN y que se pueden expresar en proteínas, a través de la información codificada en los genes. Así, la diferencia entre un mamífero, como un perro y un reptil como una lagartija, se debe a que poseen diferente información genética (diferentes genes), y la diferencia entre genes se debe a la diferencia en la secuencia en la que están ordenadas las bases nitrogenas a lo largo de cada gen. Las diferencias entre razas de perros, por ejemplo, también se deben a cambios en el ordenamiento de las bases nitrogenadas en los genes, solo que comparten más genes entre sí, lo que los hace más parecidos y sus diferencias por lo tanto a nivel de ADN son más sutiles, por lo que podemos decir que, entre más cercano sea el parentesco entre dos individuos, mayor será el grado de similitud en el ADN de ambos.
Los estudios de parentesco entre especies o entre individuos se pueden estudiar mediante distintas técnicas como veremos a continuación.
Pruebas inmunológicas
Este tipo de pruebas se pueden hacer con cualquier especie incluyendo a los vegetales, pero utilizaremos a los primates como ejemplo.
El grado de parentesco entre especies se empezó a estudiar en el nivel molecular con las pruebas inmunológicas. En 1967, Sarich y Wilson inyectaron albúmina (proteína) humana en conejos con la finalidad de obtener anticuerpos (que el organismo del conejo forma a partir de la identificación de la proteína humana como ajena al organismo y que funciona como antígeno). Los anticuerpos así formados por el conejo serán específicos contra la albúmina humana. Lo mismo se hizo con albúmina de chimpancé al inyectarla a otros conejos. Posteriormente, se extrajeron los anticuerpos para la albúmina humana y de chimpancé por separado, y se mezclaron con albúmina de otras especies y midieron el grado de reactividad. Las especies que se compararon fueron: humano, chimpancé, bonobo (chimpancé pigmeo), gorila, orangután y cercopitecos. El resultado al comparar las reacciones de las diferentes albúminas contra el anticuerpo para la albúmina humana fue: humano con un humano, la reacción fue completa, pero menor al comparar al chimpancé con el humano y todavía menor al comparar con el gorila, orangután y por último con los cercopitecos. Aunque esta prueba no implica la comparación del material genético, como la albúmina es una proteína que se produce bajo las instrucciones del ADN, esta prueba indica indirectamente y de manera somera las diferencias entre los ADN de diferentes especies.

Como podemos ver, la precisión de las reacciones inmunológicas es limitada, por lo que posteriormente a ellas se han desarrollado otras pruebas, entre ellas las de la hemoglobina, que es la molécula encargada del transporte de oxígeno en el organismo.
La hemoglobina es una proteína que está formada por dos cadenas llamadas alfa de 141 aminoácidos y dos cadenas beta de 146 aminoácidos.
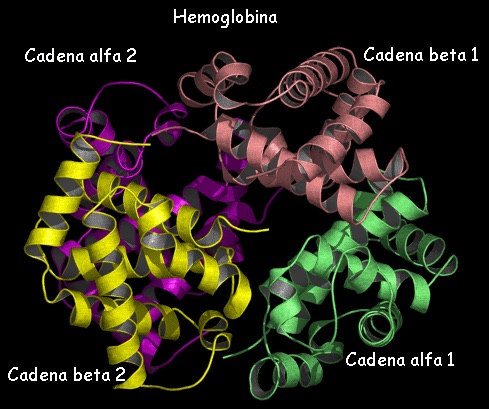
De estas, el humano y el chimpancé comparten idénticas secuencias de aminoácidos, mientras que con el gorila, el humano difiere en un aminoácido en cada cadena. En la cadena alfa, el aminoácido en la posición 23 en el humano es un ácido glutámico, mientras que en el gorila es un ácido aspártico; en la cadena beta en la posición 104 el humano tiene arginina y el gorila lisina.
El orangután y el gibón, así como otros primates, tienen también un ácido aspártico en la posición 23 de la hemoglobina alfa, lo que hace pensar que la sustitución de un ácido aspártico por glutámico tuvo lugar en el antecesor común del chimpancé y el humano (ver figur 3.)

Como las variaciones en los aminoácidos de las proteínas son resultado de cambios en el ADN, sería mejor conocer la secuencia de nucleótidos, que analizar la secuencia de aminoácidos en la proteína. Como cada triplete (codón) codifica para un determinado aminoácido, en el caso de las hemoglobinas se pueden deducir las mutaciones posibles.
Los codones del ARN mensajero que codifican para el ácido glutámico en la cadena alfa (presentes en chimpancés y humanos) son GAA y GAG y para el ácido aspártico (presente en los gorilas) GAU y GAC, el cambio de ácido glutámico por aspártico se consigue por la sustitución del tercer nucleótido en cualquiera de los tripletes posibles, mientras que los codones para la arginina (en chimpancés y humanos) son CGN (N puede ser cualquiera de las bases, ya que en la última posición de este triplete codifican para el mismo aminoácido) , AGA, AGG y lisina (en el gorila) AAA y AAG en la cadena beta. Para el primer triplete se necesitan varias sustituciones, pero para los dos últimos basta con un cambio en el nucleótido central. Esto nos lleva a la comparación en las secuencias del ADN como un método más preciso para obtener relaciones de parentesco.
La técnica más usada es la de hibridación ADN-ADN
La hibridación ADN-ADN permite comparar el grado de diferencia genética entre dos especies, ya que al separarse de un tronco común, cada una va acumulando pequeñas variaciones en su información genética y con el tiempo van siendo cada vez menos parecidas.
Existen algunas variantes para esta técnica, pero la idea principal en todas ellas es medir el grado de complementariedad entre los nucleótidos de dos cadenas de ADN diferentes.
Para realizar la técnica se requiere de pequeños fragmentos de ADN de unos 500 nucleótidos. La molécula de ADN de dos bandas es separada mediante la elevación de la temperatura, que rompe las uniones de puente de hidrógeno que mantienen unidas a las dos bandas, quedando éstas separadas. También se tienen que separar las bandas de ADN de la especie con la cual se va a comparar por medio del mismo procedimiento. Se deben mezclar ambos tipos de bandas y se deja que se unan entre sí al bajarles la temperatura (se forman nuevos puentes de hidrógeno entre nucleótidos) por lo que se aparean formando ADN híbrido, formado por una banda de cada especie. Si la secuencia de nucleótidos en ambas bandas es complementaria en su mayor parte, habrá muchas uniones entre las dos bandas, pero habrá secciones de ADN que no se complementen y éstas serán las variaciones acumuladas. Posteriormente, las moléculas híbridas se calientan para romper los puentes de hidrógeno que las mantienen unidas. Si presentan pocas uniones entre nucleótidos de ambas bandas, la temperatura a la que se separen será menor que la de una cadena híbrida que presente una mayor complementación entre ambas bandas, por lo tanto se requerirán temperaturas menores para separar bandas de especies menos relacionadas entre sí que de especies más relacionadas. El principio en el que se basa la técnica es que el ADN de especies cercanas tendrá más secuencias de nucleótidos similares que el ADN de especies con relación más distante.
Lo que se argumenta en contra de la confiabilidad de esta técnica es que sólo se analizan segmentos cortos de ADN y no el ADN completo, ya que la enorme cantidad de nucleótidos hace imposible la comparación total del ADN.
Usando esta técnica se ha podido determinar un grado de similitud entre el ADN humano y del chimpancé que ha variado entre 97, 98 y 99% (según diferentes resultados) y colocado al chimpancé como el animal más cercano al humano evolutivamente hablando, y, al chimpancé, más cercano a los humanos que a los gorilas, por lo que se ha confirmado lo que ya se sospechaba. Como se puede ver, esta técnica es una muestra de que las diferencias genéticas entre especie se deben a la separación de un tronco común. A mayor diferencia en las secuencias de nucleótidos mayor tiempo de separación de ese tronco.
Sibley y Ahlquist realizaron un amplio trabajo de hibridación de los ADN de humanos, chimpancés, gorilas, orangutánes y babuinos (Sibley y Ahlquist, J. Mol. Evol., 20,2 (1984) y encontraron gran semejanza entre chimpancés y bonobos y estos están más cercanos a los humanos que al gorila y más alejados del orangután y gibón, y todos estos más separados de los cercopitécidos. Estos estudios han sido avalados por la comparación directa de secuencias de ADN de una misma región del genoma en las especies anteriores. Esta última técnica es la que permite una mayor precisión, ya que compara secuencias específicas de ADN, dando una idea más clara del parentesco, ya que las mutaciones se van acumulando a lo largo del tiempo, y por lo tanto, entre más tiempo tengan separadas dos especies, mayor será la divergencia en las secuencias de nucleótidos
Pero ya que sabemos que las diferencias entre variedades, especies y grupos mayores se deben a cambios en el ADN ¿Qué utilidad se puede obtener de este conocimiento?. Se pueden deducir situaciones por medio de las cuales se produjeron variaciones que condujeron a divergencias evolutivas. Por ejemplo; qué relación de parentesco existe entre los humanos y los primates más cercanos y cuáles pudieron ser los cambios en el ADN que nos han hecho diferentes.
Desde hace tiempo se ha dicho que nuestro pariente más cercano es el chimpancé ya que existen evidencias que indican que compartimos cerca del 99% de la información genética.
Los humanos tenemos 46 cromosomas o 23 pares de cromosomas, mientras que el chimpancé tiene 48 o 24 pares, al igual que el gorila y orangután. Al comparar los cariotipos de ambas especies se encontró que trece pares de cromosomas son iguales y que en el humano existe la fusión de dos cromosomas (12 y 13) que ahora forman el cromosoma dos de los humanos, pero que en los chimpancés permanecen separados. Esto es lo que hace que el chimpancé tenga un par de cromosomas más que el humano.
Asao Fujiyama, del Centro de Ciencias Genómicas de Yokohama, y sus colaboradores japoneses, alemanes, chinos, taiwaneses y coreanos, miembros del Consorcio Internacional para el estudio del cromosoma del chimpancé se fijaron el objetivo de comparar el cromosoma 22 del chimpancé con su equivalente humano, que es el 21. Entre otras cosas encontraron que en los humanos existe mayor susceptibilidad a padecer SIDA, cáncer y paludismo entre otras enfermedades y que el cromosoma 21 de humanos también es causante, en el caso de un cromosoma supernumerario (trisomía 21) del síndrome de Down y de 21 enfermedades más incluyendo el Alzheimer.
Adaptación de la tabla de enfermedades causadas por un gen asociadas al brazo largo del cromosoma 21 en humanos, elaborada por el Dr. Carlos Pucharcós (Centro de Genética Médica y Molecular, IRO, Barcelona)
Los trabajos más recientes en cuanto a la comparación del genoma humano y de otros primates es el que llevó al descubrimiento del gen que al parecer permitió el desarrollo del lenguaje en los humanos. Es muy difícil llegar a conocer cómo se dio la capacidad del habla, ya que el lenguaje no deja evidencias fósiles, sin embargo, la geonómica comparada a permitido indagar el origen del lenguaje, al comparar a los genes que permiten esa capacidad en los humanos con otras especies, ya que los especialistas en genética han encontrado que los genes relacionados con el lenguaje tienen su contraparte antigua en otras especies, lo que indica que es un gen muy antiguo que ha sufrido modificaciones a lo largo del tiempo.
La historia del descubrimiento de este gen se remonta a 1990, cuando en una familia de origen paquistaní avecindada en Londres, algunos de sus miembros padecían de un desorden en la capacidad relacionada con el lenguaje, especialmente en la incapacidad para poder hablar y entender la gramática. Se les realizó un estudio que abarcó tres generaciones, en donde la mitad de los individuos estaban afectados por un trastorno que les dificultaba el movimiento de la boca, labios y lengua y que creaba problemas gramaticales, de fonética y uso del tiempo verbal adecuado.
Esta familia ahora es conocida en el mundo de la genética como la familia KE.
Según Varghan-Khadem del Instituto de Salud Infantil de Londres, en un estudio que publicó en 1995, explica que dicha incapacidad se extiende a secuencias motoras (musculares) que hacen posible la emisión de los diferentes sonidos, teniendo dificultades para cerrar los labios, abrir la boca y mover la lengua. Por medio de un “scanner” cerebral Varghan-Kahdem identificó en estos pacientes un defecto que se ubica en el ganglio basal, región en la que se interconectan el centro del lenguaje con los movimientos musculares necesarios para ello.
La misma discapacidad fue encontrada en un niño que no tenía ninguna relación de parentesco con la familia KE y, esto permitió hacer un estudio comparativo para determinar si en ambos casos la discapacidad tenía un mismo origen.
El gen mutante que se ha identificado ya en la familia KE, así como en el niño que padece el mismo desorden, está ubicado en el cromosoma 7 (región 7q31) y originalmente fue llamado SPCH1. En todos los individuos de la familia KE que presentaron la discapacidad tuvieron la misma mutación en el mismo gen, al igual que el niño no emparentado con ellos. La mutación consiste en un cambio de guanina por adenina en el exón 14 que cambia el aminoácido arginina por histidina. También se encontró que en 360 personas normales estudiadas, no tenían la mutación, por lo que se concluyó que la mutación causa los desordenes en la capacidad del lenguaje. Este gen ahora es denominado FOXP2, por pertenecer a una serie de genes llamados forkhead (“cabeza de tenedor”), llamados así por formar una pieza de la proteína que producen con esa forma) que intervienen en la transcripción del ADN a ARN, en otras palabras, son proteínas reguladoras de otros genes. En su presencia se hace la transcripción y en su ausencia no se forman las proteínas. Por lo tanto cuando existe la mutación, ésta impide la formación de ciertas proteínas que tienen relación con la capacidad de hablar.
El gene FOXP2, pertenece a una familia de genes que se pueden encontrar en animales y hongos y, que producen proteínas que regulan a otros genes, lo que les confiere un importante papel. El FOXP2 existe en mamíferos en formas ligeramente diferentes, por ejemplo, en ratones el gen es en un 93.5% igual al humano y en ellos tiene un papel importante en el desarrollo cerebral.
Si entendemos el lenguaje oral en los humanos como el empleo de palabras para expresar ideas, este debe ser entendido como un prerrequisito para el desarrollo de la cultura. El lenguaje oral humano requiere de un control fino de la laringe y la boca que no está presente en los grandes primates no humanos. Parece ser que el gen FOXP2 es el primer gen encontrado que permite la capacidad de hablar. Esto ya ha sido probado con los individuos de la familia KE que presentan alteraciones (mutaciones) en dicho gen, y se requiere de dos copias funcionales para poder hablar bien.
Wolfang et al. Secuenciaron el ADN que codifica para el gen FOXP2 en chimpancés, gorilas, orangutanes, macacos y ratones, y lo compararon con el de humano.
El gen FOXP2 codifica para una proteína de 715 aminoácidos. La diferencia entre ratones y humanos es únicamente de tres aminoácidos. Los Chimpancés, gorilas y macacos, forman proteínas idénticas, codificadas por el gen FOXP2 y tienen una diferencia con el ratón de un aminoácido dos con los humanos, mientras que el orangután tiene dos aminoácidos diferentes con los ratones y tres con los humanos.
Tan pequeños cambios en las proteínas indican un gen muy bien conservado a lo largo del tiempo, sin embargo dos de los aminoácidos diferentes entre ratones y humanos, se deben a cambios que ocurrieron después de la separación del ancestro común entre los humanos y chimpancés. Los cambios en los aminoácidos son treonina por asparagina y asparagina por serina. Cambios que se dieron en las posiciones 303 y 325 respectivamente de la proteína.
En la siguiente gráfica se pueden apreciar los cambios en los aminoácidos producto de las mutaciones y se indican los cambios “silenciosos” o neutros (modificaciones en el ADN, o mutaciones puntuales que no cambian los aminoácidos en la proteína.
Como se puede observar en la gráfica, desde que se separaron de un ancestro común, los primates y el ratón, en el chimpancé y gorila sólo han cambiado en un aminoácido en el gen FOXP2, pero desde que los humanos se separaron de los chimpancés y gorilas hace entre 4.6 y 6.2 millones de años se han producido dos cambios de aminoácidos en el linaje humano y ninguno en los chimpancés y gorilas, con la excepción de uno en el orangután, pero que lo obtuvo después de que se separó del linajes que llevó a la formación de los humanos chimpancés y gorilas.
¿Qué conclusiones podemos obtener de todo esto?. La capacidad para el lenguaje oral está en el control adecuado de la laringe y boca (control muscular) y esto lo permite el gene FOXP2 de los humanos con sus dos cambios en los aminoácidos ya mencionados. La participación del gen en el control muscular también lo hace importante en la capacidad de vocalización. Como los demás primates no presentan los cambios que los humanos tienen en el gene FOXP2 esto los incapacita para realizar los movimientos necesarios y tener el control de la laringe que les permita el habla. ¿Cómo podemos saber que son las modificaciones en el gen FOXP2 de los humanos los que le permiten articular palabras?. La respuesta está en las mutaciones existentes en algunos miembros de la familia KE, mutaciones que les impiden una comunicación oral apropiada y comprender el lenguaje y la gramática. Un solo cambio en el gen FOXP2 de una guanina por adenina, cambia el aminoácido arginina por histidina en la proteína formada y esto les confiere el desorden en el habla.
Con las mutaciones adquiridas en el gen humano FOXP2, también la laringe se amplía hacia abajo sirviendo de caja de resonancia (desafortunadamente también predispone a los humanos al atragantamiento con los alimentos). Estos cambios fueron seleccionados positivamente. Esto lo podemos notar al observar que al separarse los humanos de los chimpancés los dos cambios se encuentran en todos los individuos humanos (excepto los mutantes como en la familia KE) sin importar su origen (se han realizado estudios del gen FOXP2 en individuos de muy diferentes orígenes y regiones geográficas y en todos se encuentra que forman la misma proteína), pero ninguno de los demás primates presentan estos cambios. La razón de una selección natural a favor de los cambios en el gen FOXP2 de los humanos, es que es más fácil sobrevivir en un grupo, teniendo un medio de comunicación que no sólo les permita comunicarse entre contemporáneos en un mismo lugar y tiempo, sino también transmitir ideas y conocimientos a través de generaciones, llevando esto a la aparición de la cultura.
Desde luego, el pensar que un sólo gen sea el responsable del lenguaje oral no es totalmente aceptado ya que se requiere también de cerebro complejo, de las uniones necesarias entre neuronas y el pensamiento abstracto, pero sin las capacidades oral-facial que permite el gen FOXP2, sería imposible el lenguaje oral.
Independientemente de lo anterior, lo más importante es que nos damos cuenta de como los cambios a nivel molecular, desde los muy notorios hasta los más sutiles, pueden influir en la diversidad genética en nuestro planeta.
Bibliografía.
Enard, Wolfang et al. Molecular evolutio of FOXP2, a gene involved in speech and language. Nature 418, 869 –872 (2002)
Fortna A, Kim Y, MacLaren E, Marshall K, Hahn G, et al. (2004) Lineage-Specific Gene Duplication and Loss in Human and Great Ape Evolution. PloS Biol 2(7): e207 DOI:10.1371/ journal.pbio.0020207.
Lai et al. A forkhead-domain gene is mutated in a severe speech and language disorder. Nature 413, 519 –523 (2001)
Tattersall, Ian. How we came to be Human. Scientific American. Special edition. Vol 16, Number 2, 66 –73 (2006)
Vargha-Khadem et al. Praxiic and nonverbal cognitive deficits in a large family with a genetically transmitted speech and language disorder. Proc Nat Acad Sci USA 92,930 –933 (1995)
